FOCUS PLATEFORME : Protéomique-Gif (I2BC): des analyses de spectrométrie de masse performantes à l’exploration en profondeur du protéome
La platefome Protéomique-Gif (SICaPS) de l’Institut de Biologie Intégrative de la Cellule (I2BC, Gif-sur-Yvette, Institut Joliot, CEA, Saclay), propose un ensemble d’analyses protéomiques dédiées à l’analyse en profondeur et la comparaison de protéomes, d’interactomes et de modifications post-traductionnelles. Parmi ses équipements de spectrométrie de masse, le timsTOF Pro de Bruker (acquis grâce à des financements SESAME Santé IdF, Plan Cancer, IBiSA, Université Paris-Saclay et CNRS) permet d'identifier plusieurs milliers de protéines à partir de petites quantités d’extraits protéiques totaux (50 à 200 ng de protéines). Combinée à des méthodes de préparation des échantillons adaptées et à des analyses statistiques multivariées innovantes, la technologie timsTOF Pro offre une profondeur d’analyse qui permet d’étudier finement l'expression normale ou altérée des protéines de différentes souches en fonction de conditions biologiques telles que la composition du milieu de culture, la durée de la culture ou autre paramètre clé. Ces approches offrent des perspectives prometteuses pour accéder à une compréhension globale/systémique des processus moléculaires qui sous-tendent un fonctionnement cellulaire normal ou altéré/pathologique.
Qu’est-ce que la technologie timsTOF Pro ? Dans le spectromètre de masse timsTOF Pro, la technologie « Trapped Ion Mobility Spectrometry » (TIMS) permet de séparer et concentrer par mobilité ionique les ions de même rapport masse-sur-charge mais de conformations différentes. Combiné à un analyseur hybride composé d’un quadripôle de sélection ultra-rapide et un analyseur à temps-de-vol, d’excellente résolution, il permet d’acquérir les spectres de fragmentation en mode PASEF (Parallel Accumulation & Serial Fragmentation) avec des vitesses d’acquisition MS/MS, sensibilités et sélectivités élevées. Alors qu’une analyse protéomique classique de type bottom-up, par nanoLC-MS/MS, permet de caractériser chaque peptide protéolytique par son temps de rétention en chromatographie liquide de phase inverse, son rapport masse-sur-charge et son spectre de fragmentation MS/MS, la séparation par mobilité ionique dans la cellule TIMS permet d’augmenter encore la sélectivité des analyses ce qui ajoute une quatrième dimension aux analyses protéomiques. Ainsi le timsTOF Pro permet de réaliser des analyses protéomiques 4D et d’identifier et quantifier un grand nombre de protéines, même peu abondantes, à partir d’échantillons complexes.
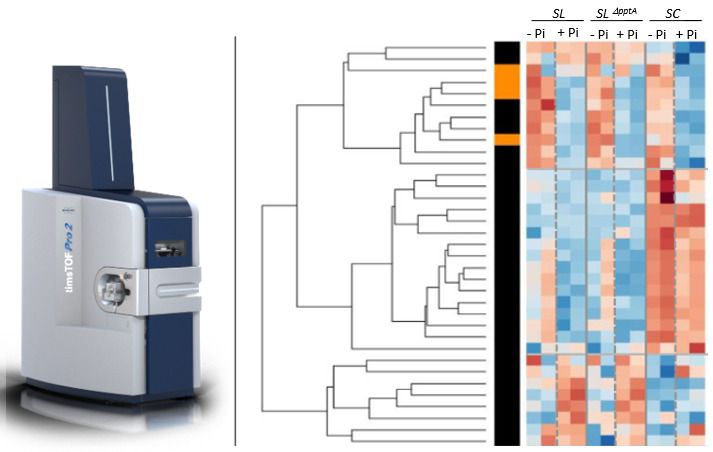
Un exemple d’application ? Regard sur le protéome de Streptomyces et l’analyse en profondeur d’un très grand jeu de données aux multiples variables ! Dans le cadre d’un projet collaboratif avec l’équipe « Métabolisme Energétique des Streptomyces » (MES) de l’I2BC dirigée par Marie-Joelle Virolle (marie-joelle.virolle@i2bc.paris-saclay.fr), le timsTOF Pro a permis de comparer les protéomes de 3 souches de Streptomyces avec une combinaison de variables (souches sauvages ou mutantes, temps de culture, milieu de culture carencé ou non en phosphate). Chez les Streptomyces, la biosynthèse des antibiotiques est déclenchée en condition de limitation nutritionnelle en phosphate qui est généralement corrélée à un déficit en ATP (stress énergétique). Deux souches de Streptomyces, S. coelicolor et S. lividans, phylogénétiquement très proches, présentent des capacités très différentes à produire les mêmes métabolites spécialisés bio-actifs (antibiotiques). Dans cette étude, les différentes conditions et variables ont été analysées en quadruplicats pour un total de 48 échantillons. La profondeur de l’analyse protéomique réalisée en mode Data-Dependent Acquisition sur le système nanoLC-timsTOF Pro a permis d’identifier et quantifier plus de 4000 protéines de Streptomyces, soit plus de la moitié des protéines prédites in silico. Au-delà de l’analyse sensible par spectrométrie de masse, c’est l’analyse statistique de l’énorme jeu de données protéomiques comportant de nombreuses variables (type de souches, temps et milieu de culture, etc..) qui a été un des principaux défis pour aboutir à une analyse en profondeur, robuste et fiable, des données protéomiques de quantification relatives entre les réplicats de plusieurs conditions expérimentales. L’exploitation de ces données quantitatives multivariées et corrélées a ainsi été rendue possible par le développement d’une approche statistique reposant en partie sur l’utilisation d’un modèle linéaire généralisé à effets fixes (souches, temps et milieu de culture). D’une manière intéressante, la richesse des données générées et ainsi analysées a permis d’établir le profil métabolique et physiologique d’une souche fortement productrice d’antibiotiques (S. coelicolor), par rapport à une souche phylogénétiquement très proche mais faiblement productrice des mêmes antibiotiques (S. lividans).
En savoir plus ? Cette étude protéomique combinant les performances du spectromètre de masse timsTOF Pro à une analyse statistique multivariée innovante des données a permis de générer une quantité inédite d’information et de mener une analyse protéomique comparative en profondeur de trois souches de Streptomyces, en fonction de la disponibilité en phosphate, en collaboration avec l’équipe de Marie-Joelle Virolle. L’ensemble des résultats a donné lieu à 3 publications : Lejeune et al., Production Int J Mol Sci 2022 ; Lejeune et al., Front Microbiol 2022 ; Lejeune et al., Res Microbiol 2024.
Cette étude illustre l’intérêt et le potentiel des approches de protéomique ultra sensibles en profondeur sur des protéomes totaux et l’expertise de la plateforme Protéomique-Gif non seulement pour l’obtention de données de protéomiques de qualité mais aussi pour l’adaptation de l’analyse des données de protéomiques à des plans expérimentaux complexes. Les nouvelles performances du timsTOF Pro a ainsi permis d’accéder à une profondeur d’analyse des protéomes jamais atteinte et l’arrivée prochaine d’un spectromètre de masse encore plus sensible devrait permettre d'accroitre encore la profondeur des analyses.
Si les analyses de protéomes ou d’interactomes complexes vous intéressent, n’hésitez pas à nous contacter et à nous soumettre vos demandes. Nous élaborerons ensemble la stratégie la plus adaptée à votre projet.
-> Contacts : Laila Sago (laila.sago@i2bc.paris-saclay.fr), David Cornu (david.cornu@i2bc.paris-saclay.fr) et Virginie Redeker (virginie.redeker@cnrs.fr)
Plug In Labs Université Paris-Saclay : cliquer ICI
Aussi, en 2021, la plateforme publiait son premier FOCUS PLATEFORME. Le relire ? FOCUS PLATEFORME : Protéomique-Gif (SICaPS) : une plateforme dédiée à l'exploration des protéomes et de leurs modifications (27-sept.-21)
Protéomique-Gif (SICaPS), La plateforme Protéomique-Gif (SICaPS), labellisée IBiSA, est ouverte à l'ensemble de la communauté scientifique. La plateforme propose des méthodologies de protéomique et des technologies de spectrométrie de masse de pointe pour identifier, caractériser et quantifier les protéines et leurs modifications à partir d'échantillons plus ou moins complexe. Une technologie de dernière génération permet l'identification et la quantification relative de protéines peu abondantes dans des échantillons protéiques très complexes. Des méthodes analytiques adaptées permettent de caractériser des modifications co- , post-traductionnelles ou chimiques des protéines (phosphorylations, lipides, pontages covalents, …). La plateforme dispose d'une solide expertise dans la préparation d'échantillons protéiques de nature variée, leur analyse par spectrométrie de masse et le traitement des données. Elle optimise et développe des méthodes analytiques pour réponse aux besoins des utilisateurs. La plateforme propose une variété de services sous forme de prestations : i) standards : préparations d'échantillons, mesures de masse exacte, identification de protéines, contrôle-qualités de protéines recombinantes ; ii) collaboratives : quantification relative des protéines, comparaisons approfondies de protéomes, interactomes, caractérisation de déterminants structuraux et de modifications des protéines.
A propos de l’Institut de Biologie Intégrative de la Cellule (I2BC - UMR 9198). L’I2BC est une Unité Mixte de Recherche (CEA, CNRS, Université Paris-Saclay), accueillant une soixantaine d’équipes de recherche et hébergeant 17 plateformes technologiques, réparties en 6 pôles. 2025 est aussi une année clé pour l’I2BC : cette unité fête ses 10 ans cette année.